刘若尘博士为第一作者,裴炎教授为通讯作者,在国际期刊《New Phytologist》发表了题为《DNA methylation occurring in Cre‐expressing cells inhibits loxP recombination and silences loxP‐sandwiched genes》的研究论文,2021年该期刊影响因子为10.051。研究揭示了位点特异性重组酶系统在植物中删除效率低下的原因,为改进重组酶系统的效率提供了理论依据;阐明在一些高效的花特异启动子控制下,重组酶可以介导大片段序列高水平的DNA甲基化和多基因沉默,为研究DNA甲基化起始机制和开发新型高效定点DNA甲基化工具提供了新模型和新策略。

本研究在烟草中筛选一系列启动子,研究了影响位点特异性DNA重组反应效率的因素。发现:(1)早期花特异启动子驱动下的Cre/loxP系统可以使靶标基因产物(mRNA和蛋白质)完全消除,然而靶标基因(DNA)却并未被真正地删除;(2)在Cre的作用下,loxP位点和位点间的靶标基因发生高度DNA甲基化修饰,导致基因删除被抑制和靶标基因沉默;(3)loxP位点上重组酶结合区域的CG甲基化对基因删除没有影响,而交叉区域的CHH(H = A、T或C)甲基化在抑制loxP重组反应中起关键作用,致使Cre/loxP基因删除系统在富含非-CG甲基化背景的植物中效率低下;(4)其它重组酶系统,包括Flp/FRT、KD/KDRT、和B3/B3RT也有类似地低基因删除效率和高DNA甲基化的现象。
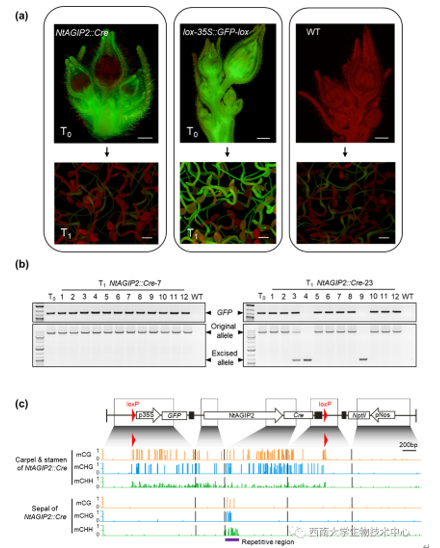
图2.1 重组酶Cre介导的DNA甲基化抑制loxP重组同时沉默loxP位点间的基因